Detecting selection in low-coverage high-throughput sequencing data using principal component analysis, BMC Bioinformatics
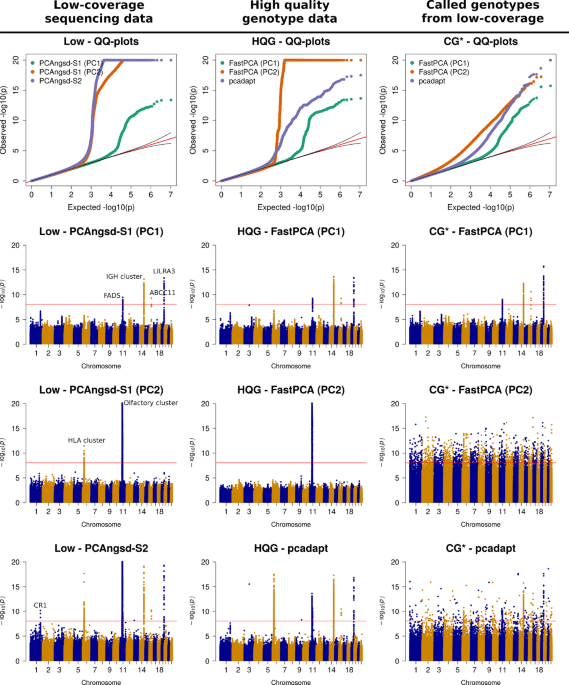
Background Identification of selection signatures between populations is often an important part of a population genetic study. Leveraging high-throughput DNA sequencing larger sample sizes of populations with similar ancestries has become increasingly common. This has led to the need of methods capable of identifying signals of selection in populations with a continuous cline of genetic differentiation. Individuals from continuous populations are inherently challenging to group into meaningful units which is why existing methods rely on principal components analysis for inference of the selection signals. These existing methods require called genotypes as input which is problematic for studies based on low-coverage sequencing data. Materials and methods We have extended two principal component analysis based selection statistics to genotype likelihood data and applied them to low-coverage sequencing data from the 1000 Genomes Project for populations with European and East Asian ancestry to detect signals of selection in samples with continuous population structure. Results Here, we present two selections statistics which we have implemented in the PCAngsd framework. These methods account for genotype uncertainty, opening for the opportunity to conduct selection scans in continuous populations from low and/or variable coverage sequencing data. To illustrate their use, we applied the methods to low-coverage sequencing data from human populations of East Asian and European ancestries and show that the implemented selection statistics can control the false positive rate and that they identify the same signatures of selection from low-coverage sequencing data as state-of-the-art software using high quality called genotypes. Conclusion We show that selection scans of low-coverage sequencing data of populations with similar ancestry perform on par with that obtained from high quality genotype data. Moreover, we demonstrate that PCAngsd outperform selection statistics obtained from called genotypes from low-coverage sequencing data without the need for ad-hoc filtering.

Next generation sequencing technologies to explore the diversity

SCALA: A complete solution for multimodal analysis of single-cell

PDF) Robust principal component analysis for accurate outlier

A novel workflow for the qualitative analysis of DNA methylation

Region-specific denoising identifies spatial co-expression
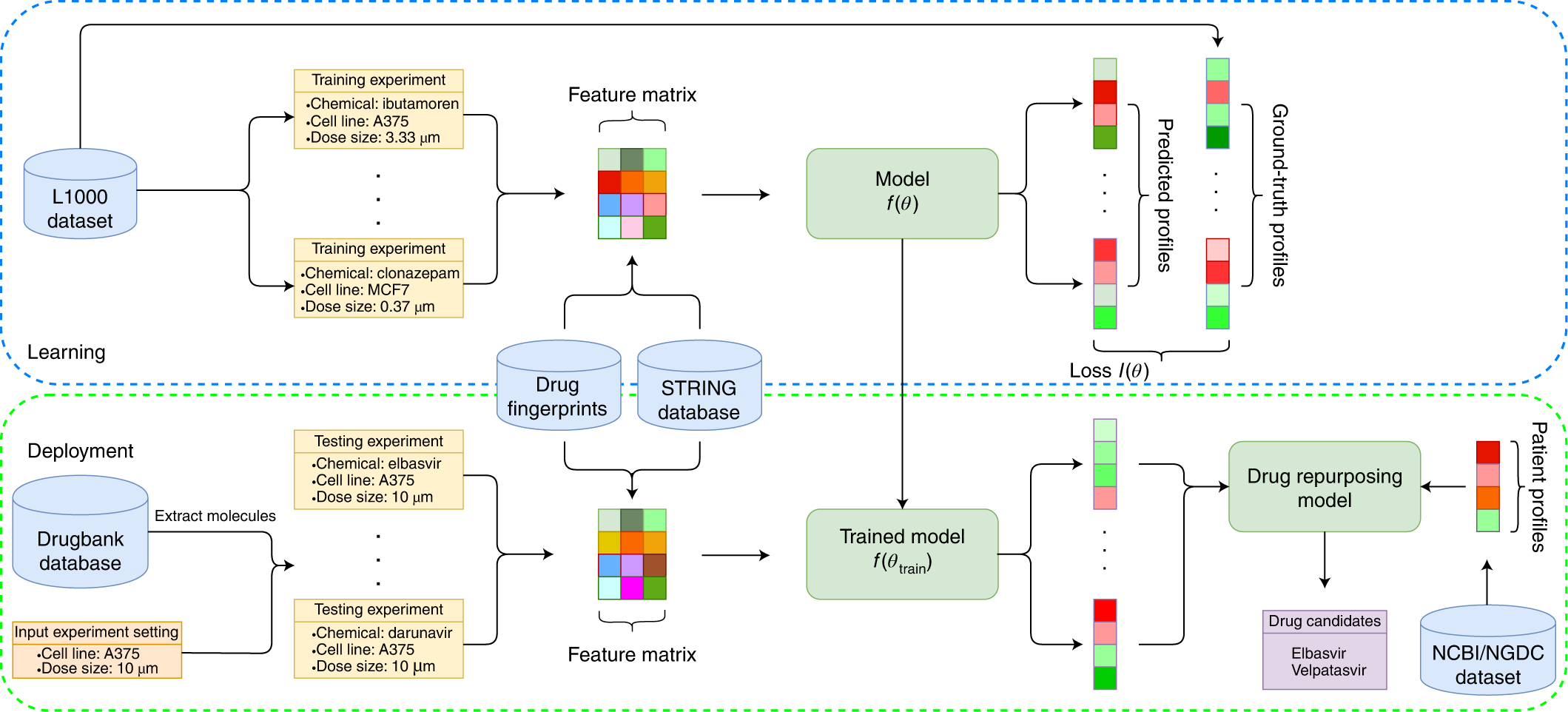
A deep learning framework for high-throughput mechanism-driven

Chromosomal inversions harbour excess mutational load in the coral, Acropora kenti, on the Great Barrier Reef

Generation II DNA Sequencing Technologies - Drug Discovery World (DDW)
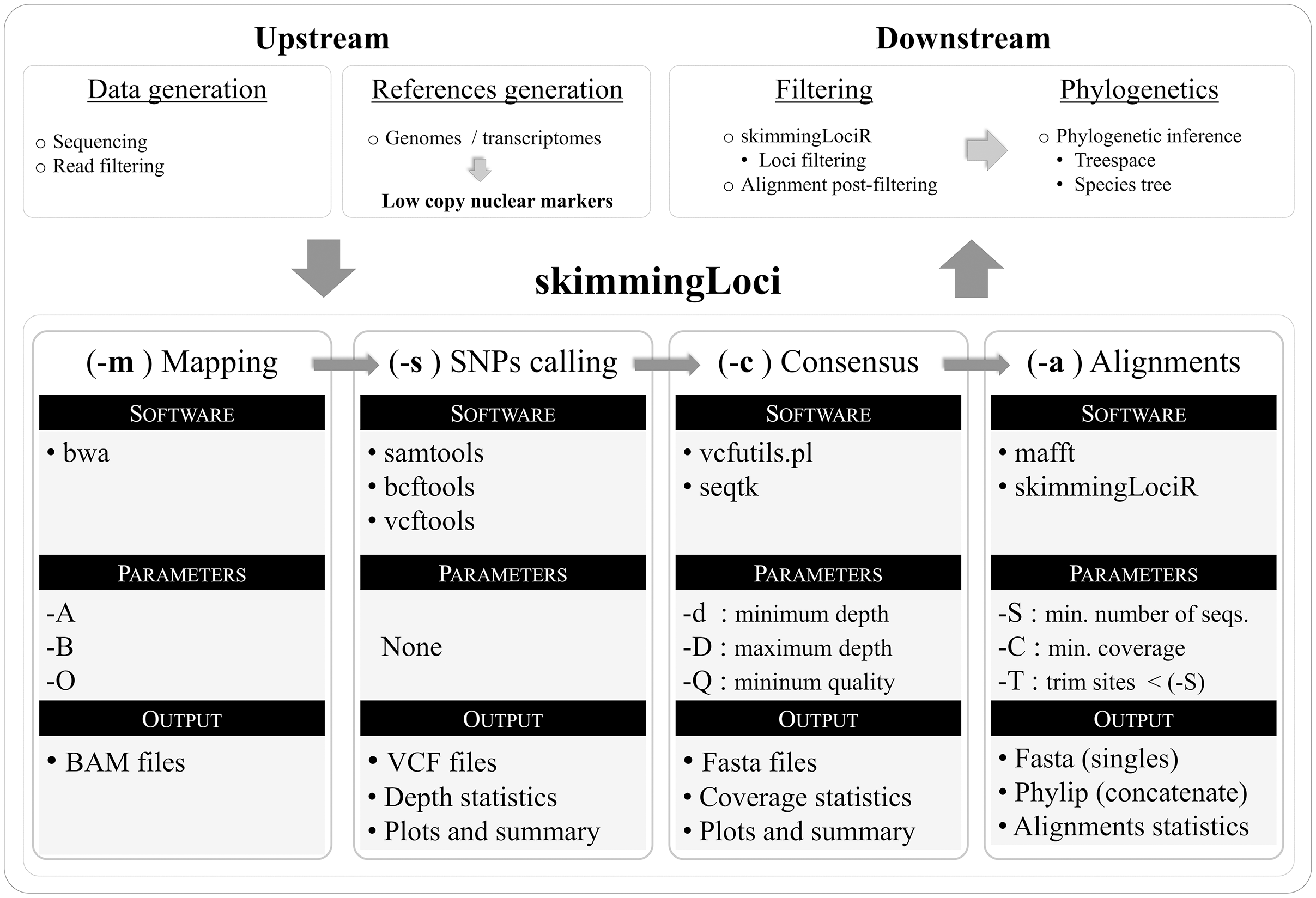
A pipeline for assembling low copy nuclear markers from plant
Selection hypotheses and their encodings as covariance matrices